Enginyeria genètica: Aquest migdia, la Reial Acadèmia Sueca de Ciències comunicava la decisió d’atorgar el Premi Nobel de Química d’enguany conjuntament a Emmanuelle Charpentier i a Jennifer A. Doudna “pel desenvolupament d’un mètode d’edició genòmica”. Es refereixen al mètode d’edició genètica pel sistema de CRISPR-Cas9, que ha obert els darrers anys una nova perspectiva en l’enginyeria genètica, car és una tècnica més específica, més dirigida i més eficient en la substitució d’informació genètica en sistemes biològics.

Esquema de l’edició genètica pel sistema CRISPR-Cas9
Emmanuelle Charpentier

Emmanuelle Charpentier (*Juvisy-sur-Orge, 11.12.1968) estudià bioquímica, microbiologia i genètica a la Universitat Pierre i Marie Curie, de París. Realitzà la tesi doctoral de l’Institut Pasteur sobre mecanismes moleculars de la resistència bacteriana a antibiòtics (1995). El 1996 passà al laboratori d’Elaine Tuomanen (*1951), on estudià els elements genètics mòbils responsables de l’alteració genòmica de Streptococcus pneumoniae. El 1997 treballà en el laboratori de Pamela Cowin al New York University Medical Center sobre la regulació del creixement del pèl en ratolins. El 2002 encapçalà el seu propi laboratori a la Universitat de Viena, dedicat a l’estudi de la regulació de la virulència de Streptococcus pyrogenes. El 2009 es traslladà a la Universitat d’Umeå, i el 2014 a Centre Helmholtz de Recerca d’Infeccions de Braunschweig. Des del 2018 és directora de la Unitat Max Planck de Ciència de Patògens, de Berlin.
Jennifer A. Doudna

Jennifer Anne Doudna (*Washington, D.C., 19.2.1964) estudià bioquímica al Pomona College de Claremont (California), on es graduà el 1985. Realitzà la tesi doctoral a la Harvard Medical School sobre un sistema per augmentar l’eficiència d’un ribozim autoreplicatiu (1989). El 1994 passà a Yale, on treballà sobre el ribozim de grup I de Tetrahymena i sobre el ribozim del virus de l’hepatitis D. El 2002 es traslladà a la UC Berkeley. El 2009 començà a treballar per Genentech, però aviat ho deixà per tornar a Berkeley amb una recerca sobre les potencialitats del sistema CRISPR. Actualment compagina la plaça de professora a Berkeley amb la d’investigadora del Howard Hughes Medical Institute.
Un mètode d’edició genètica basat en el sistema CRISPR-Cas9
Ishino et al. (1987) en investigar la seqüència nucleotídica del gen iap d’Escherichia coli remarcà la presència d’una repetició consisteix en cinc seqüències altament homòlogues de 29 parells de bases (pb), amb una simetria diàdica de 14 pb i separadors de 32 pb. Mojica et al. (1993) detectaren una estructura semblant en el genoma d’Haloferax mediterranei, un arqueó halofílic: 14 seqüències gairebé perfectament conservades de 30 pb repetides a distàncies regulars. Fou el grup de Francisco J. M. Mojica, de la Universitat d’Alacant, qui estudià la rellevància biològica d’aquestes estructures i el fet que hom les trobés de manera prou comuna en els genomes de bacteris, arqueons i mitocondris (Mojica et al., 2002). Jansen et al. (2002) es referí a aquesta família de seqüències amb l’acrònim CRISPR (repeticions palindròmiques breus interespaiades regularment en agregats). Associat a les CRISPR apareixen un grup de gens, que Jansen et al. designaren com a “cas” (CRISPR-associated). Els gens cas, de fet, eren exclusius de posicions adjacents a CRISPRs. Els productes gènics de “cas” presentaven motius d’helicasa i nucleasa, la qual cosa feia pensar que eren proteïnes reguladores de l’expressió gènica o del metabolisme general de l’ADN. L’acumulació de dades genòmiques i el desenvolupament de la bioinformàtica permeté la descripció d’un gremi de 45 famílies “cas”, amb els corresponents subtipus de CRISPR/Cas (Haft et al. 2005).
Mojica et al. (2005) trobaren que les seqüències CRISPR deriven d’elements genètics aliens, com ara bacteriòfags o plàsmids. El grup de Mojica i d’altres arribaren a la conclusió que la incorporació de seqüències CRISPR constituïa un mecanisme de protecció per a bacteris i arqueons front a la infecció per bacteriòfags o la transfecció per plàsmids. Aquest mecanisme de protecció és de caràcter específic, i es comporta com una autèntica memòria d’agressions genètiques del passat.
Les CRISPRs del genoma són transcrites en llargues molècules d’ARN (pre-crRNA) que són tallades per generar molècules més petites (crRNAs; CRIPSPR-related RNAs). Les seqüències de crRNA poden reconèixer seqüències homòlogues en genomes forans (fags o plàsmids) introduïts a la cèl·lula i destruir-los. En aquest procés de reconeixement i destrucció participen les proteïnes Cas.
Barrangou et al. (2007) confirmaren les sospites d’altres grups: el sistema CRISPR forneix a bacteris i arqueons un mecanisme de resistència adquirida contra virus. Hom ha vist una analogia amb el sistema d’ARN d’interferència dels organismes eucariòtics i, de manera més llunyana, amb el sistema immunitari específic de vertebrats.
El grup d’Emmanuelle Charpentier es dedicà a l’estudi dels mecanismes de maduració de crRNA en Streptococcus pyogenes, determinant el rol que hi jugaven la RNasa III i petites molècules d’ARN (tracrRNA) codificades en llocs distants del genoma (Deltcheva et al., 2011). En aquest procés també participava la proteïna Cas9, que actuaria d’àncora entre les tracrRNA i el pre-crRNA.
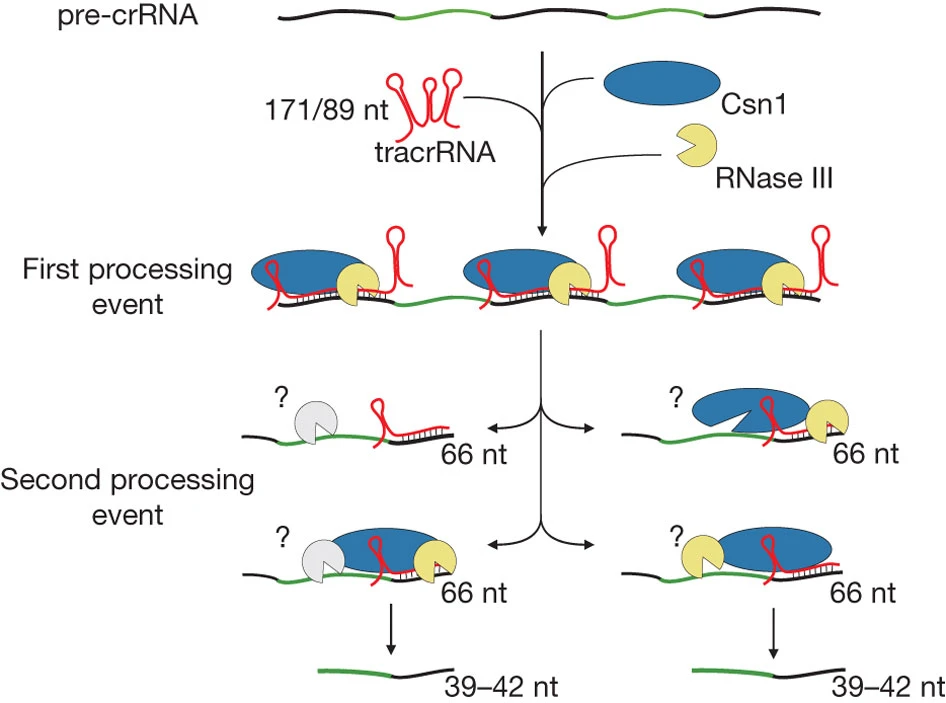
Model de maduració del pre-crRNA (pre-CRISPR-related RNA) proposat pel grup de Charpentier en el 2011
El grup de Charpentier col·laborà amb el de Jennifer A. Doudna per estudiar si el crRNA podia ser utilitzat per dirigir l’especificitat de seqüència de l’activitat nucleasa. Trobaren que, si més no en alguns d’aquestes sistemes, el crRNA forma amb el tracrRNA una estructura comuna que dirigeix la Cas9 a la introducció de talls en la seqüència diana d’ADN (Jinek et al., 2012). D’aquesta manera, la tracrRNA no tan sols desencadena la maduració de pre-crRNA a crRNA a través de la RNasa III, sinó que també activa l’atac de la Cas9 a la seqüència corresponent d’ADN. La Cas9 té aquesta activitat ADNasa a través de dos dominis HNH i RuvC, cadascun dels quals talla una de les cadenes de l’ADN bicatenari.
Doudna i Charpentier tenien en ment les possibilitats d’aquest sistema en l’enginyeria genètica. Per això sintetitzaren una molècula d’ARN de guia simple (sgRNA) a partir de la fusió de crRNA i tracrRNA. D’altra banda, observaren que podien alterar la seqüència diana si alteraven la seqüència de sgRNA. El sistema sgRNA/Cas9 tenia, doncs, la possibilitat de tallar de manera específica qualsevol seqüència d’ADN, és a dir de fer edició genòmica programable per ARN. Ben aviat es demostrà que aquest sistema CRISPR/Cas9 tenia realment la capacitat de fer edició genètica en línies cel·lulars humanes (Mali et al., 2013).
Lligams:
- Pressmeddelande: Nobelpriset i kemi 2020.
- CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Elitza Deltcheva, Krzysztof Chylinski, Cynthia M. Sharma, Karine Gonzales, Yanjie Chao, Zaid A. Pirzada, Maria R. Eckert, Jörg Vogel & Emmanuelle Charpentier. Nature 471: 602-607 (2011).
- A Programmable Dual-RNA–Guided DNA Endonuclease in Adaptive Bacterial Immunity. Martin Jinek, Krzysztof Chylinski, Ines Fonfara, Michael Hauer, Jennifer A. Doudna, Emmanuelle Charpentier. Science 337: 816-821 (2012)
Cap comentari:
Publica un comentari a l'entrada